Contribución:
Lic. Geog. Natalia Raptópulos
Infraestructura de Datos Espaciales (IDECOR)
17 de junio 2020.
Hay una serie de herramientas libres para procesamiento de imágenes que vale la pena conocer y sacar provecho. Entre ellas podemos mencionar Orfeo Toolbox, Google Earth Engine (GEE), SOPI de Conae y SNAP de la ESA.
En esta nota explicaremos paso a paso, cómo elaborar el Índice de Vegetación Normalizado (NDVI) utilizando el software SNAP.
¿Qué son los índices espectrales?
A partir de la información de las distintas bandas de las imágenes satelitales, se pueden construir numerosos índices espectrales.
Los índices espectrales son realces que consisten en adjudicar a cada píxel un valor que dependerá exclusivamente del valor que el mismo tenga en dos o más bandas espectrales.
Dichos índices espectrales o estandarizados, nos permiten identificar ciertos elementos o comportamientos de la superficie terrestre.
Uno de los más conocidos es el NDVI o Índice de Vegetación Normalizado, que se utiliza en gran medida para el monitoreo de la vegetación, ya que es un buen estimador de la actividad fotosintética y, por lo tanto, permite distinguir rápidamente la vegetación en una imagen satelital.
Software SNAP
Para calcular el NDVI, en este caso, utilizaremos el software SNAP. SNAP (Sentinel Application Platform) es un programa gratuito proporcionado por la ESA (Agencia Espacial Europea).
Fue desarrollado especialmente para el análisis de imágenes de satélites provenientes, principalmente, de su flota de satélites Sentinels, pero también se pueden trabajar imágenes provenientes de otros satélites, ya que admite numerosos formatos.

Les compartimos el link para descargar el programa. En este caso, utilizamos la última versión disponible: 7.0.0
Paso a paso: Cómo calcular NDVI
El proceso para elaborar índices espectrales con el software SNAP, es verdaderamente muy simple y rápido.
En este caso, trabajamos sobre una imagen Sentinel 2A, obtenida el día 20 de marzo de 2020. La imagen abarca una zona del Departamento San Martín, donde se destaca la ciudad de Villa María al Sureste.

Estas imágenes, las pueden descargar de distintos portales de descarga como Copernicus Open Access Hub de la Esa o EarthExplorer del USGS.
Una vez cargada la imagen en SNAP, recomendamos los siguientes pasos:
1) Resampling o Remuestreo:
Para poder realizar operaciones entre bandas es necesario que todas las bandas posean la misma resolución espacial. Por esto, realizamos un resampling o remuestreo.
La función Resampling es un algoritmo que permite otorgar a las bandas que se seleccionen, la resolución geométrica que se desee. Para el caso de Sentinel 2A, se suele remuestrear en 10m, 20m o 60m, ya que sus bandas tienen estas distintas resoluciones.
Para realizar esto ir a:
- Barra de herramientas principal: Raster – Geometric operations – Resampling
- En la pestaña I/O Parameters de la ventana Resampling, en la opción Source Product seleccionar la imagen que se desea remuestrear.
- En la opción Target product aparecerá por defecto el nombre del archivo de salida del proceso, el cual será igual al del archivo de origen, con el sufijo “_resampled”.
- En la Opción Save As se puede elegir el formato de salida y en Directory se debe buscar el directorio donde será guardada la imagen.
- Si no se cuenta con buena cantidad de memoria de procesamiento les recomendamos en esta etapa, guardar la imagen en formato BEAM-DIMAP, formato original de SNAP que se procesa más rápido y utiliza menos recursos computacionales. Luego se puede exportar al formato que se desee.
- En la pestaña Resampling Parameters se colocan los parámetros según el tipo de análisis que se desea realizar, en este caso lo pasaremos a 10 m de resolución espacial. El programa propone diferentes opciones para el remuestreo. En este caso, se seleccionó la opción por banda (By reference band from source product), escogiendo la banda 2 (de 10 metros de resolución) como referencia.
- En método de remuestreo (Upsampling method), el SNAP propone tres modalidades: Nearest, Bilinear y Bicubic. En este caso se elige la opción Nearest y por ultimo ejecutar el proceso.
Hay que tener en cuenta que las imágenes Sentinel ocupan mucho espacio en disco por lo que estos procesos suelen demorar bastante tiempo. Si la zona de interés es más chica que la imagen total, se puede realizar un recorte previo.
Luego de terminado el proceso, aparecerá el nuevo archivo generado, en el explorador de productos (a la izquierda de la pantalla).
2) Índices espectrales:
Como dijimos al principio, elaborar índices espectrales con SNAP es muy simple y rápido.
Simplemente debemos dirigirnos a Optical – Thematic Land Proccesing y allí elegir qué tipo de variable queremos observar en la superficie. Por ejemplo, para el caso del NDVI, seleccionarlo dentro de la categoría de índices de vegetación (Vegetation Radiometric Indices – NDVI/Processor).
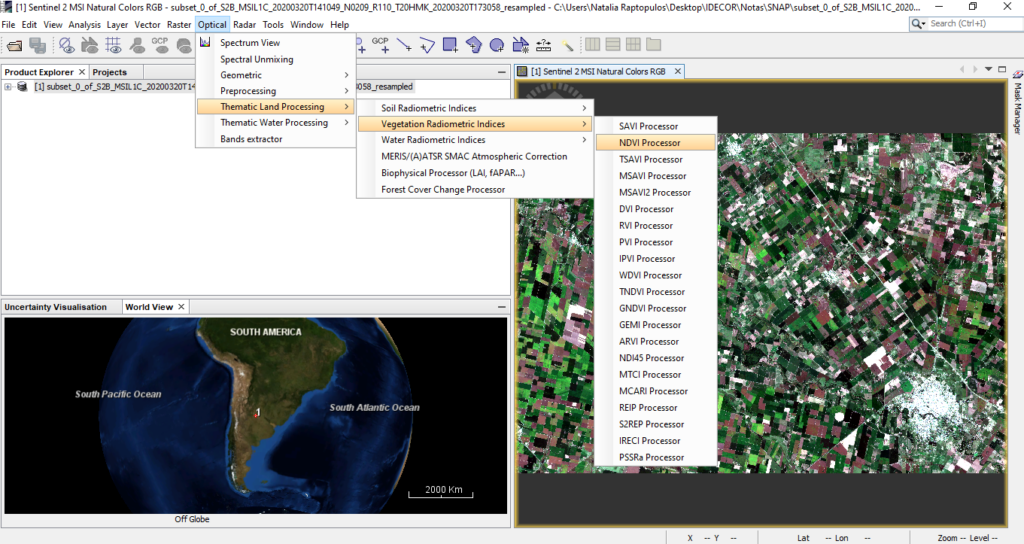
Luego, se abrirá una ventana donde debemos seleccionar en la pestaña I/O Parameters la imagen remuestreada y en “name” colocar el nombre y debajo la ubicación del nuevo producto que se generará.
En la solapa “Processing Parameters” debemos indicar qué banda utilizaremos para calcular este índice. En este caso, para calcular NDVI solicita ingresar la banda Roja e Infrarroja, ya que la fórmula para el cálculo de este índice, es la siguiente:
NDVI = (IRC – R) / (IRC + R)
Para el caso de Sentinel 2A, como banda roja utilizamos la banda 4 y la banda 8 para el infrarrojo cercano o NIR. Hacemos click en Run y se lleva a cabo el proceso.
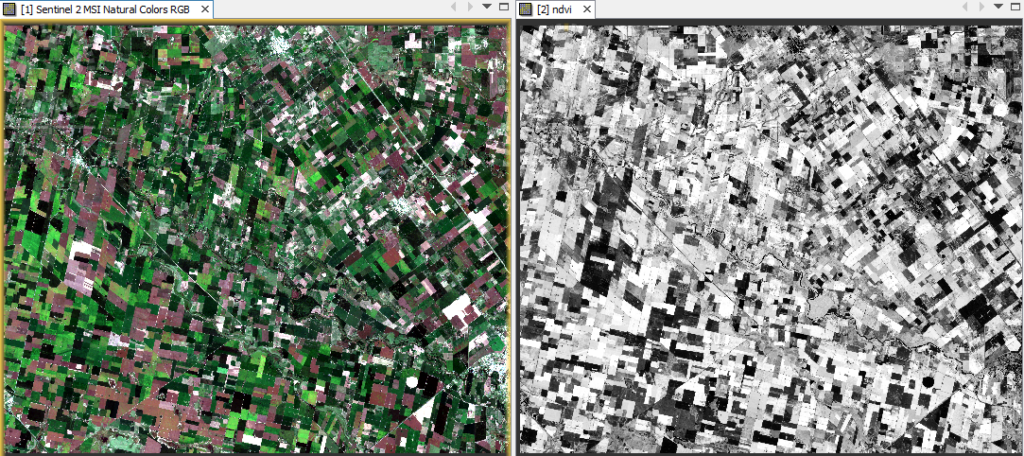
Rápidamente se generará el nuevo producto, que aparecerá en la ventana de “Product Explorer”, y si nos fijamos en las bandas, figura el NDVI generado sobre el cual haremos doble clic para visualizarlo.
Así, obtendremos una imagen en escala de grises, donde los valores obtenidos varían entre -1 y 1. Mientras más cercano a 1 indica presencia de vegetación, cercano a 0 muy probablemente nos indique suelo desnudo y si es negativo, en general nos indica presencia de agua.
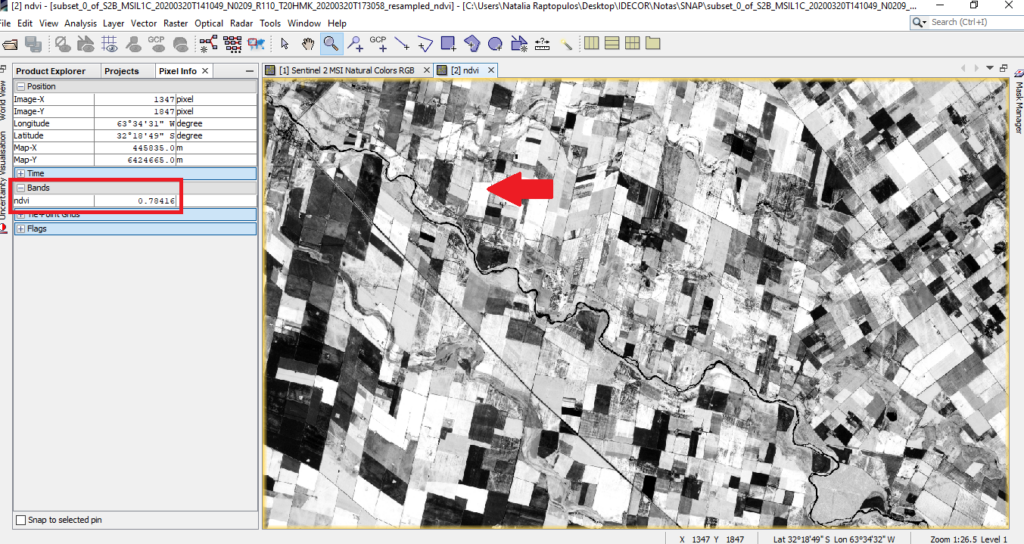
Finalmente, podemos ir a la solapa de Píxel info, y al recorrer la imagen, ir viendo los valores de NDVI. Donde los colores más claros nos indican valores más altos y los más oscuros más bajos. Este mayor valor nos indica presencia de vegetación con mayor actividad fotosintética o más vigorosa y los valores más bajos pueden indicar vegetación con menor densidad, enferma o senescente, y como dijimos antes, valores cercanos a cero son áreas de suelo desnudo o sin vegetación, y valores negativos nos indicarían presencia de agua.
¿Cómo exportamos nuestra imagen de NDVI?
El software SNAP nos brinda la posibilidad de exportar lo generado en múltiples formatos compatibles con la mayoría de los softwares de procesamiento de imágenes satelitales y SIG.
Para realizar esto, en primer lugar hay que seleccionar la imagen a exportar y luego debemos ir a Archivo – Exportar y elegir el formato de salida. Por ejemplo, “GeoTIFF” es un formato ampliamente aceptado por diversos softwares, y permite conservar la información georreferenciada, los datos de proyección y valores de cada banda, entre otras cosas. Luego indicamos el nombre y dónde guardar el nuevo archivo generado y… listo!
Como vimos, este mismo proceso podemos realizarlo para elaborar numerosos índices ya que SNAP tiene gran variedad, y también tenemos la posibilidad de construir índices propios.
¿Qué otros procesos te gustaría aprender a realizar en SNAP? Envianos tus sugerencias a nuestro mail [email protected].
Y recordá que podés suscribrte a nuestro portal, accediendo al siguiente link.




